研究成果
研究成果
第49回NMR討論会で発表しました
NMR試料管内転写によるRNAのNMRスペクトルの測定
斉藤裕之1,伊谷野悠里1,牛田千里2,清澤秀孔3,○河合剛太1
(1千葉工大・工,2弘前大農生, 3遺伝研・新領域融合研究セ)
- Introduction
Expression of small RNA, with 50-100 nt length, from the locus of the sense-antisense transcript in mouse genome was found and investigation of the function of such small RNA is being conducted. In order to characterize the structures of the small RNAs from mouse, a method of in tube transcription is introduced.
- SAT遺伝子座における低分子RNA出現の例
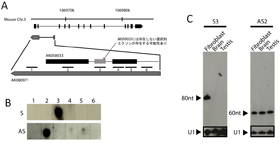
Fig. 1 Expression of small RNA form a SAT locus.
(A) A set of transcripts and positions of DNA probes. (B) Hybridization with sense and antisense probes. Strong hybridizations were observed for two probes, sense-3 (S3) and antisense-2 (AS2). (C) S3 probe hybridized on RNA with a length of about 80nt in a tissue specific manner. AS2 probe hybridized on RNA with a length of about 60nt without tissue specificity.
右の図の領域では、DNAの両鎖からの発現が確認されている。このように両鎖からの発現が見られる領域をSAT(sense-antisense transcript)遺伝子座と呼んでいる。このSAT遺伝子座の特定の領域に対応する配列を持つ低分子RNAが発見されている。
マウスにおいて、センス・アンチセンス発現が確認されている遺伝子座は4000を超えており、その多くの遺伝子座で低分子RNAが発現していることが示唆されている。
なお、ここではセンス鎖はデータベースに登録された鎖を指し、タンパク質のコーディングの有無とは無関係である。
Okada, Y., et al., Hum. Mol. Genet. 17, 1631-1640 (2008)
- NMR試料管内転写によるRNAの構造スクリーニング
マウスの多数のSAT遺伝子座に由来する低分子RNAが見つかっており、一方、次世代シーケンサーによるマウス由来RNAの直接解析によって、100万を超える低分子RNAの塩基配列が得られている。これらの解析によって得られる機能性RNAが立体構造を形成するかどうかを迅速に解析するために、NMR試料管内での転写を試みた。
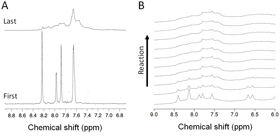
(A) 13C-selected spectra. In the beginning of the reaction, strong and sharp signals due to the labeled NTPs were observed. After the 2 days measurements, signals only from the transcript were observed.
(B) Time course of normal spectra.
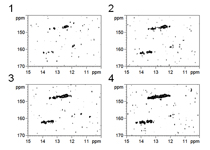
Fig. 3 15N-1H HMQC spectra
Imino proton signals from the base paired residues were increased during the reaction.
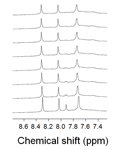
Fig. 4 13C-selected spectra
The reaction was not completed due to the wrong preparation of NTPs (Bad example).
- Methods
The reaction mixture was prepared in a microtube and then transferred into the Shigemi NMR tube. NMR measurements were performed with a 800 MHz spectrometer equipped with a cryoprobe (bruker) at a probe temperature of 298 K.
- S3 RNAの二次構造解析
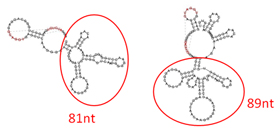
Fig. 5 Prediction of secondary structure of the S3 RNA
Two possible structures were predicted by the program vsfold (http://www.rna.it-chiba.ac.jp/vsfold5/). Pseudoknot formation was also predicted as shown in red.
SAT遺伝子座に由来するS3 RNAが特定の二次構造を形成するかどうかを調べるため、二次構造予測およびNMRスペクトルの測定を行った。
S3 RNAは、40残基のDNAプローブによるノーザン解析によって80残基程度であることが分かっているが、正確な長さ(あるいは配列)はわかっていない。プローブの前後の領域を含めた二次構造予測の結果、二次構造を形成する領域の候補として、Fig. 5に示す2つの可能性が示唆された。
そこで、2つのRNA候補に対応する鋳型DNAを設計し、MR試料管内転写法によってイミノプロトンスペクトルを測定した。その結果、81残基のRNAについては、良く分散した比較的シャープなシグナルが得られたことから、安定な二次構造および高次構造の形成が推定された。
- NMR試料管内転写法の利点
1.反応に必要なものを混合し、NMR試料管に入れるだけ.鋳型DNAおよび合成したRNAの精製のステップを省略。
2.15N標識NTPを用いれば、合成されたRNAのイミノプロトンシグナルのみを観測できる。
3.13C標識NTPを用いることにより、さらに多くの情報を得ることができると考えられる。
4.転写反応の進行状況も確認できる。
- Results and discussion
To determined the exact sequence of the S3 RNA, secondary structure of the RNA with upstream and downstream sequences was analyzed. By using the in tube transcription method, formation of stable secondary structure was found for one of the two candidates of S3 RNA (the 81nt RNA). Thus, the 81nt RNA is possibly the functional unit of the S3 RNA.
- Conclution
The in tube transcription method can be a powerful tool to analyze the large number of structured small RNA candidates.
本研究は、新学術領域研究「マウス細胞内に普遍的に存在する50-100ヌクレオチド低分子RNAの多角的解析」の一環として行われた。また、先端研究施設共用促進事業による「RNAの構造スクリーニングおよび構造クラスタリング手法の開発」として、理研SSBCのNMR分光計を利用した。